DISEÑO DE BIOFÁRMACOS: UNA ALTERNATIVA EN LA BÚSQUEDA DE TRATAMIENTOS PARA COVID-19
Carlos Yabar 1
Resumen: El avance de la investigación en el campo de la biología molecular y la bioinformática ha permitido que la simulación de prototipos en biofármacos se presenten como una opción promisoria para buscar potenciales tratamientos para diferentes enfermedades transmisibles y no transmisibles. En el siguiente artículo se describen los principales hallazgos en la investigación de los biofármacos con énfasis en SARSCOV2.
Palabras clave: Biología molecular, bioinformática, biofármacos.
Introducción
La biología molecular en armonía con otras disciplinas está facilitando el diseño y desarrollo de biofármacos, muchos de los cuales ya están en fases avanzadas. Se trata de insumos biológicos producidos para fines terapéuticos, profilácticos y de diagnóstico, los cuales son generados mediante el uso de la biotecnología (1). Asimismo, en la era post-genómica, también se han desarrollado de manera importante plataformas de bioinformática, lo que ha permitido complementar los análisis proteómicos y de interacción de moléculas que podrían ayudar a entender cómo un biofármaco es capaz de interaccionar con una proteína que proviene de un agente infeccioso de interés (2).
En tal sentido, la biología molecular y la bioinformática, han logrado establecer un binomio perfecto para la comprensión de la interacción molecular entre las proteínas virales y del hospedero. Ejemplo de ello, es la investigación enfocada al modelaje de proteínas por homología, la cual consiste en identificar la secuencia de un gen de interés, traducir la secuencia de nucléotidos a aminoácidos y seguidamente, realizar la construcción de la figura tridimensional de la proteína con base a una proteína homóloga ya cristalizada y modelada previamente (estudios de proteómica).
Ensayos in silico
Este proceso de modelado molecular, permite simular la interacción con otras moléculas según su afinidad estructural o química, proceso conocido como “acoplamiento molecular” del inglés docking (3). A través de este análisis, Lu et al., (4) revelaron que el subdominio externo de la proteína S de SARS-CoV2 presentaba una alta homología estructural con el subdominio del SARS-CoV del año 2002 y, por tanto, su afinidad era alta frente al receptor de la enzima convertidora de la angiotensina 2 (ACE2 por sus siglas en inglés). Este hallazgo fue importante para subsecuentes estudios dirigidos a la interacción de estas dos proteínas. Con relación a ello, una investigación logró modelar péptidos in silico (expresión que significa hecho por computadora o vía simulación computacional), diseñados a partir de la cadena alfa-hélice del receptor ACE2, los cuales presentaron una fuerte afinidad electroestática por el dominio de unión del receptor de la proteína S del virus, convirtiéndose en fuertes candidatos a fármacos contra el SARS-CoV2 (5).
Asimismo, la interacción simulada de la proteína de envoltura viral E con diferentes fitoquímicos, identificó tres moléculas: Belachinal, Macaflavanona E y Vibsanol B que evitaban el movimiento aleatorio de la proteína viral E en su calidad de canal iónico transmembrana, sugiriendo propiedades inhibitorias de estos fitoquímicos (6). Las propiedades de fármacos ya identificados como efectivos en otros virus han sido también evaluados in silico con proteínas modeladas de SARS-CoV2. Un estudio demostró que algunos inhibidores de proteasa del virus de la hepatitis C, se unía con gran afinidad con el sitio activo de la proteasa Mpro según el score de análisis docking, sugiriendo que podría tratarse de un inhibidor potencial contra esta proteasa viral (7). Del mismo modo, el nelfinavir, un potente inhibidor nucleósido de la transcriptasa reversa de VIH, también presentó una fuerte interacción con la hélice de la región HR1 de la proteína S in silico, implicada en procesos de fusión de membrana, inhibiendo la formación de sincitio celular in vitro (8). Si bien, su actividad farmacológica no ha sido probada en humanos con SARS-CoV2, sigue siendo considerado en la lista de posibles fármacos contra COVID-19 (9).
Biotecnología de anticuerpos
Si bien, la terapia con plasma convaleciente permite disponer de anticuerpos humanos específicos contra el SARS-CoV2 favoreciendo la rápida recuperación en pacientes críticos (10,11), este procedimiento requiere también la búsqueda de pacientes voluntarios recuperados para abastecer de suficiente plasma a través del tiempo. A ello se suman los procedimientos estándar para su procesamiento, y medidas óptimas de bioseguridad para evitar la presencia de partículas extrañas, alérgenos y patógenos (10).
Una novedosa alternativa frente a esta limitación ha sido el desarrollo del sistema Phage Display, el cual permite sintetizar anticuerpos recombinantes humanos a través de una “fábrica” biológica constituida por Escherichia coli y un bacteriófago (12). Ello permite que, durante el proceso de infección del virus en la bacteria, se vaya sintetizando el anticuerpo humano, cuyo gen que lo codifica se encuentra insertado en la nucleocápside viral. En ese sentido, la síntesis del anticuerpo se incrementa a medida que se va replicando el bacteriófago en E coli (Figura 1).
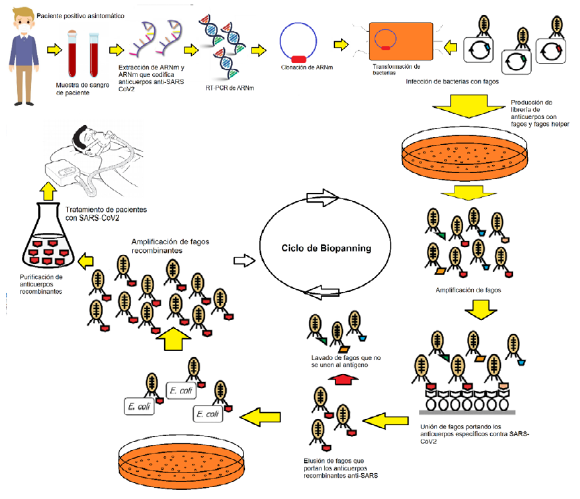
La aplicación de esta novedosa herramienta fue orientada en sus inicios hacia la inmunoterapia contra enfermedades hematológicas, autoinmunes, en desórdenes neurológicos, como trombocitopenia aloinmune neonatal y tumores cancerígenos (13). Sin embargo, su aplicación se ha extendido hacia el tratamiento de enfermedades infecciosas causadas por algunos virus, bacterias hongos y parásitos, entre los que destaca el virus SARS-CoV de la pandemia de 2002, donde a través de la fusión del anticuerpo con la proteína de la espícula, se logró evitar la unión del virus con el receptor de la célula en ensayos in vitro (12,14).
Conclusión
En conclusión, los biofármacos se presentan como una propuesta importante de medicamentos basados en técnicas de simulación basados en biología molecular y bioinformática, los cuales podrían ser útiles para cubrir la falta de antivirales efectivos y específicos contra el SARS-CoV2. Para ello se requiere su evaluación por las diferentes etapas y fases experimentales de ensayos clínicos.
Referencia bibliográfica
1. Matar, P. Biofármacos y Biosimilares – Riesgos y desafíos en los nuevos desarrollos. Hematología. 2008; 12(2): 57-59
2. Cai Y, González JV, Liu Z, Huang T. Computational systems biology methods in molecular biology, chemistry biology, molecular biomedicine, and biopharmacy. Biomed Res Int. 2014:746814. doi: 10.1155/2014/746814.
3. Barradas-Bautista D, Rosell M, Pallara C, Fernández-Recio J. Structural Prediction of Protein-Protein Interactions by Docking: Application to Biomedical Problems. Adv Protein Chem Struct Biol. 2018; 110:203-249. doi: 10.1016/bs.apcsb.2017.06.003.
4. Lu R, Zhao X, Li J, Niu P, Yang B, Wu H et al. Genomic characterization and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020;395(10224):565-574. doi: 10.1016/S0140-6736(20)30251-8.
5. Han Y, Král P. Computational Design of ACE2-Based Peptide Inhibitors of SARS-CoV-2. ACS Nano. 2020:acsnano.0c02857. doi: 10.1021/acsnano.0c02857.
6. Gupta MK, Vemula S, Donde R, Gouda G, Behera L, Vadde R. In-silico approaches to detect inhibitors of the human severe acute respiratory syndrome coronavirus envelope protein ion channel. J Biomol Struct Dyn. 2020 Apr 15:1-11. doi: 10.1080/07391102.2020.1751300.
7. Bafna K, Krug RM, Montelione GT. Structural Similarity of SARS-CoV2 Mpro and HCV NS3/4A Proteases Suggests New Approaches for Identifying Existing Drugs Useful as COVID-19 Therapeutics. ChemRxiv [Preprint]. 2020 Apr 21. doi: 10.26434/chemrxiv.12153615.
8. Musarrat F , Chouljenko V, Dahal A, Nabi R, Chouljenko T, Jois SD et al. The anti‐HIV drug nelfinavir mesylate (Viracept) is a potent inhibitor of cell fusion caused by the SARSCoV‐2 spike (S) glycoprotein warranting further evaluation as an antiviral against COVID‐19 infections. J Med Virol. 2020;1–9. doi: 10.1002/jmv.25985
9. Barlow A, Landolf KM, Barlow B, Yeung SYA, Heavner JJ, Claassen CW et al. Review of Emerging Pharmacotherapy for the Treatment of Coronavirus Disease 2019. Pharmacotherapy. 2020;40(5):416-437. doi: 10.1002/phar.2398.
10. Rajendran K, Krishnasamy N, Rangarajan J, Rathinam J, Natarajan M, Ramachandran A. Convalescent plasma transfusion for the treatment of COVID-19: Systematic review. J Med Virol. 2020 May 1. doi: 10.1002/jmv.25961.
11. Shen C, Wang Z, Zhao F, Yang Y, Li J, Yuan J et al. Treatment of 5 Critically Ill Patients With COVID-19 With Convalescent Plasma. JAMA. 2020;323(16):1582–9. doi: 10.1001/jama.2020.4783.
12. Chan SK, Rahumatullah A, Lai JY, Lim TS. Naïve Human Antibody Libraries for Infectious Diseases. Adv Exp Med Biol. 2017;1053:35-59. doi: 10.1007/978-3-319-72077-7_3.
13. Bazan J, Całkosinski I, Gamian A. Phage display-a powerful technique for immunotherapy: 2. Vaccine delivery. Hum Vaccin Immunother. 2012 Dec 1;8(12):1829-35. doi: 10.4161/hv.21704. Epub 2012 Aug 21.
14. Jin Y, Lei C, Hu D, Dimitrov DS, Ying T. Human monoclonal antibodies as candidate therapeutics against emerging viruses. Front Med. 2017; 11(4):462-470. doi: 10.1007/s11684-017-0596-6.
Citar como: Yabar C. Diseño de biofármacos: Una alternativa en la búsqueda de tratamientos para COVID-19. Bol Inst Nac Salud. 2020; 26 (11-12):140-142.
1 Centro Nacional de Salud Pública- Instituto Nacional de Salud, Lima, Perú.










